TOP > ニュース・特集 > QBiCのこだわり > 生命動態データベース統合化で加速するデータ駆動型生物学
生命動態データベース統合化で加速するデータ駆動型生物学

生命科学は大規模データの時代に突入した
一昔前の家庭用のパソコンでは、ワープロやお絵描きソフトを使うのがやっとだったが、最近のパソコンでは動画の再生や制作も軽々とできるようになった。こうしたコンピュータの性能の向上は大型コンピュータを使う研究の世界も大きく変えつつある。神戸の多細胞システム形成研究センター(旧発生・再生科学総合研究センター)内に研究室を構えるQBiC発生動態研究チーム(大浪修一チームリーダー)では、小さなモデル動物を使って、その発生の詳細を大量の動画データとして記録している。
大学院生の時から大浪の研究室に所属し、それ以来、生物学と情報処理の両方に関わってきた京田耕司研究員が見せてくれたのは、コンピュータ画面に大写しにされた小さな生きものの卵だった。 C.elegans とよばれる体長1mmほどの線虫の受精卵だ。1970年代には一つの受精卵が卵割してから、約1,000個の細胞でできた成虫になるまで、すべての細胞分裂が記録されたという。当時は人間が顕微鏡で観察しながら、細胞の分裂の様子を手書きのメモとして記録していった。現在は顕微鏡とコンピュータをつなげて自動で大量の断層写真を撮影しハードディスクに記録していく。それらの断層写真を重ね合わせることによって三次元(3D)画像が再構成される。さらに一定時間ごとに3D画像を取得することによって細胞分裂という現象の時間変化を詳細に記録していくことができる。こうしたシステムを4D顕微鏡と呼んでいる。
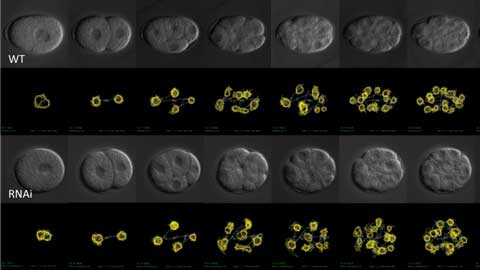
4D顕微鏡によって撮影した線虫C. elegansの細胞分裂の時系列画像(左から右へ)と、そこから画像解析によって抽出してきた核の位置と動き(黄色線)。正常な線虫(WT、上半分)と、RNAiという方法によって特定の遺伝子を阻害した線虫(下半分)について比較した。
京田らは4D顕微鏡を使って、遺伝子機能を阻害したさまざまな胚(発生の初期段階にある個体)について細胞分裂の詳細な解析を行なっている(図1)。コンピュータの画面の上に映し出され、時々刻々変化する細胞の様子をコンピュータが画像処理して細胞核の輪郭やその分裂の様子を数値情報として抽出してくる。主観の入る余地のない高精度なデータが得られ、かつその取得の速度も格段に上がった。
大規模解析が一般的になって生物学のデータ蓄積のスピードはますます加速している。大規模データベースには様々なものがある。ゲノム情報、マイクロアレイを使ったトランスクリプトーム情報、プロテオーム情報、メタボローム情報。いずれも物質の構造や頻度に関する網羅的大規模データベースだ。これらのデータは生物のある状態の平均値を表しているといえよう。一方、QBiCが注目しているのは個々の要素の時空間変化を包括するデータである。たとえば生きた細胞の分裂や形態変化の進行にともなう分子や細胞内小器官の組成や局在の変化。動物の発生過程や動物の行動を時間を追って記録したデータなど。また様々なコンピュータシミュレーションからも要素の時空間変化を含むデータが得られる。こうしたデータを解析し、細胞の挙動や生命現象の予測を目指す研究が始まりつつある。世界的にもこうしたデータベースの構築とそれを利用した研究分野が盛んになっていくと考えられる。
データベースの共有と利用の問題点

有用なデータベースは多数公開されているが実際にアクセスするのは難しい。
科学研究の発展のためには情報の共有が必須だ。一人の研究者が生み出せるデータ量には限りがある。情報共有のメリットは、他の研究者のデータを自由に入手し、それらを比較解析できることだ。しかし、そこには大きな障壁があると京田研究員は言う。
「研究者は皆、独自に研究プロジェクトを行なっています。そしてデータの形式はプロジェクトごとに決められているのが普通です。つまり、二つの研究グループが似たような研究プロジェクトを行なっていても、全く違うデータ形式を採用しているということもありえます。そうなると、お互いのデータを直接比較することは難しくなります。」
実験科学者は自分の研究にとって使い勝手のよい道具を選ぶ傾向にある。京田自身、長年、細胞分裂の時空間の動態を定量計測するプロジェクトとそれらのデータベース構築に関わってきたが、そこでは一つの細胞の核を表現するデータ形式として線分を繋いで記述された多角形の集合を採用している。一方、アメリカのグループは核を表現する形式として球体を使っている。
「データ形式が異なることでいろいろな問題に突き当たります。公開されている有名なデータベースの中には、中身を見ることさえ簡単ではないものがあるんですよ。」
データはハードディスクに記録された数字の列であるため、それを解釈し、人の目に見えるようにするためにはソフトウェア(可視化ソフト)が必要だ。しかしデータベースの中には汎用の可視化ソフトが認識できないデータ形式が使われているものが多い。それぞれのデータ形式専用の可視化ソフトを使えば見ることはできるが、比較解析をするときに同じソフトウェアを使えないというのははなはだ都合が悪い(図2)。
すべての研究者が使えるデータ形式をつくる
QBiCを含め世界の多くの研究所でシステム生物学的アプローチを用いて、生命現象を予測しようとする研究が行われている。そうした研究では要素の時空間における動態を記述したデータが蓄積されている。これらのデータベースはどれもまだ歴史が浅いため共通規格化はなされていなかった。京田らは主要なデータベースを構築している研究者たちと連携し、世界ではじめて標準データフォーマット策定に着手し、BDML(Biological Dynamics Markup Language)という形式にまとめあげ発表した[1]。
データ形式の標準化のポイントの一つ目の特徴はXML(Extensible Markup Language)を基盤とする形式を採用したことだ。XML形式とは、テキスト情報を基本として、すべての情報を情報の意味や構造を表すタグで囲む形式だ。その利点はコンピュータにとってだけでなく、人間にとってもデータの意味が理解しやすいことだ。XML形式は、たとえばPubMed などの文献情報データベースなどにも利用されているほか、様々なアプリケーション間の情報のやりとりに利用されている。また、ウェブサイトを構成する文書などもXML形式から派生したものが使われている。
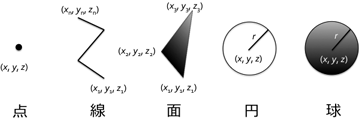
二つ目の特徴は空間的情報の表現を規格化すること。BDMLでは最小エレメントとして、点、連続した線分、三角形(面を表現)、円、球体が用意されている(図3)。これらの要素を組み合わせると既存のほぼすべてのデータ形式に対応できる。もちろん、XMLには拡張性があるので将来必要に応じて新たなエレメントを加えることも可能だ。
三つ目の特徴はデータの中身ではなく、データの由来や実験条件などについての情報、いわゆるメタデータを検索可能な形で格納できるようにしたことだ。メタデータに含まれる情報にもとづいて由来の異なるデータ同士の関連性を知ることができる。そのためにはメタデータの表現をコンピュータによって判別可能な形式(主語、述語、目的語の3個の組み合わせだけで表現する)に規格化する必要がある。さらにあいまいさを回避するために、すべての用語についてオントロジー(特定の分野で使われる概念を体系的に整理し、データベース化したもの。生物学に関しては遺伝子オントロジーというものがある)を利用してIDをつけるという工夫がなされている。
BDMLで記述されたデータはQBiC発生動態研究チームが管理するSystems Science of Biological Dynamics (SSBD) データベース(http://
最近の細胞レベルのモデル化技術の発展により、細胞骨格や細胞小器官の動態などがシミュレーションできる時代になった。BDML形式を用いることで、画像から定量化されたデータのみならず、細胞レベルのシミュレーションの結果も記述することが可能である。これによって実験から得られる計測データとシミュレーションの結果を比較・解析することも可能になる。
生命科学の未来:解析ツールの共有化と機械学習
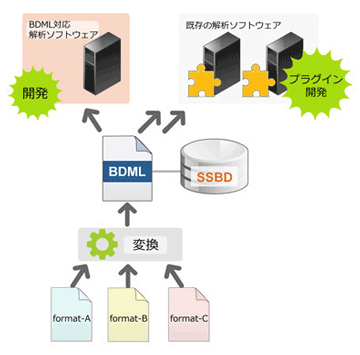
データ解析ソフトには様々なものがある。しかし現在データベースを超えて共通に使えるものはほとんどない。BDML形式を扱える解析ソフトを開発すればBDMLに準拠したすべてのデータベースのデータを解析できる。そのメリットは計り知れない。京田らはまず、汎用の可視化ソフトや既存の代表的解析ソフトのためのプラグインを開発しBDML形式のデータを扱えるようにした。またBDMLに対応した独自の解析ソフトを開発し、SSBD データベース上で公開している(http://
「ここまではいわば長い遠回り」と共著者のひとり遠里由佳子研究員は言う。彼女はもともと大学院で人工知能の研究をしていたが、一旦一般企業に就職してから、ふたたび大規模データ解析やシミュレーションによる生物学の研究の世界へ戻ってきた。現在、システム開発の経験が豊富なホー・ケネスと協力してSSBDデータベースの設計に関わりつつ、独自の解析ソフトの開発も手がけている。
遠里が目指しているのは機械学習を応用した新しい解析ソフトの開発だ。BDML標準化が進めば、多種多様なデータを一度に扱えるようになる。種を超えたデータの比較や、分子レベル、細胞レベル、個体レベルとレベルを超えたデータを同時に扱うこともできるようになるだろう。これらの膨大なデータの中から意味を見いだすことは、もはや人間の直感だけでは不可能だ。そこに機械学習を取り入れていくのは必然的流れだろう。
大量のデータからいかにして意味ある情報を読みとるのか?この問題を解決しない限り、現代の生物学は宝の持ち腐れ状態に陥る。生物の理論的理解はこれまで数理モデルの構築とシミュレーションを中心に行われてきた。今後は実験に基づいた大規模データをコンピュータや人工知能で解析し、そこから法則性を見いだすことが可能になるだろう。こうしたアプローチを大浪はデータ駆動型(data-driven)生物学と呼び、今後のトレンドになると考えている。
- Kyoda K, Tohsato Y, Ho KH, Onami S (2015) Biological Dynamics Markup Language (BDML): an open format for representing quantitative biological dynamics data. Bioinformatics, 31(7),1044-1052 doi:
10.1093/ bioinformatics/ btu767


